基础医学院鲁林荣团队《Nat.Methods》合作发表开发图谱级数据整合分析工具scAtlasVAE并构建泛疾病人类CD8+T细胞图谱
来源:浙江大学 时间:2024-12-18 12:43:42
CD8+ T细胞在适应性免疫应答中扮演着重要角色,它们通过T细胞受体(TCR)精准识别与感染、炎症及癌症的相关抗原,从而消除外界抗原【1-2】。然而,值得注意的是,CD8+ T细胞在不同的生理和病理条件下,展现出了极为显著的表型异质性【3】。近年来,单细胞免疫分析技术的迅猛发展为我们带来了海量的单细胞T细胞数据集以及与之配对的TCR信息【4】。为了全面深入地理解CD8+ T细胞在免疫系统中的多样化作用,将这些数据集进行整合并据此构建一个人类CD8+ T细胞的综合性参考图谱,已成为一项至关重要的任务。
近日,浙江大学基础医学院鲁林荣团队与浙江大学爱丁堡大学联合学院刘琬璐团队以及腾讯AI Lab合作在Nature Methods杂志发表了文章,题为Integrative mapping of human CD8+ T cells in inflammation and cancer。同期,团队也在Nature Methods发表题为Human CD8+ T cell map with single-cell transcriptome and TCRαβ information的Research Briefings。
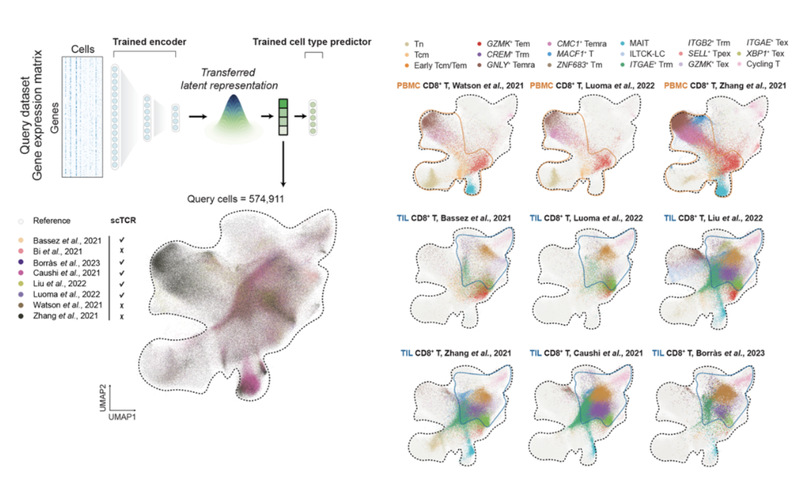
该研究开发了适用于图谱级别scRNA-seq数据的单细胞整合方法scAtlasVAE。利用该方法,研究团队建立了涵盖1,151,678个细胞的泛疾病人类CD8+ T细胞图谱。这些细胞来源于961个样本、68项研究,涵盖了42种不同的疾病状况,并配有完整的TCR信息。这一图谱不仅全面揭示了CD8+ T细胞的异质性,更为深入理解其在各种疾病情境中的作用提供了有力的支持。
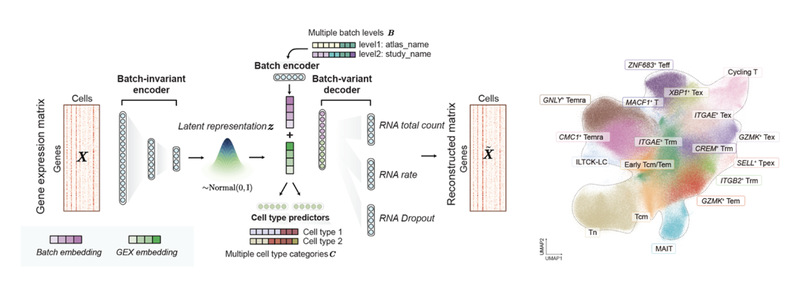
利用这一参考图谱,该研究对癌症和炎症中人类CD8+ T细胞的表型异质性进行了全面且深入的分析。通过整合TCR克隆扩增和共享信息,我们揭示了不同亚型之间的潜在联系,并发现了它们可能存在的表型和功能转变。例如,我们的方法成功表征了三种不同的耗竭CD8+ T细胞亚型。这些亚型在癌症、自身免疫性炎症以及免疫相关不良事件(irAEs)中展现出了独特的转录组特征和克隆共享模式。
此研究的泛疾病人类CD8+ T细胞图谱与scAtlasVAE模型共同为基础科学家和临床研究人员提供了宝贵的资源。当与其他参考图谱相结合时,scAtlasVAE更成为了研究人员进行图谱规模scRNA-seq数据整合和跨图谱比较的强大工具。由于我们的图谱整合了来自多种疾病状况的CD8+ T细胞数据,因此在功能相关性和普遍性之间取得了较好的平衡。
展望未来,揭示TCRαβ库的功能多样性将对于进一步理解CD8+ T细胞的功能异质性及其与各种疾病的关联具有至关重要的作用。同时,对CD4+ T细胞的深入分析、单细胞水平的T细胞跨物种比较,以及整合T细胞与其他细胞类型之间的空间相互作用,都将为我们更加深入地理解T细胞在生理和病理条件下的功能异质性提供新的视角和思路。
浙江大学医学院鲁林荣教授,浙江大学爱丁堡大学联合学院刘琬璐研究员,腾讯AI Lab医疗首席科学家姚建华为本文通讯作者,浙江大学基础医学院博士研究生吴利则,浙江大学爱丁堡大学联合学院博士研究生薛子为,为本文共同第一作者,腾讯AI Lab何冰、赵宇研究员,ZJE2021级生物信息学专业本科生赵炳康、李易诚也共同参与此课题,该项目也收到浙江大学基础医学院汪洌教授的大力支持,项目获得了国家自然科学基金、腾讯AI Lab犀牛鸟专项研究计划的支持。
鲁林荣团队主要从事T细胞生物学与自身免疫性疾病发病机制研究。在既往研究中揭示了胸腺T细胞阳性选择中TCR信号增敏新机制,发现Th细胞分化调控新途径,并率先将CAR-T技术应用到自身免疫性疾病治疗。







